针对地市级千万级人口的核酸检测系统,相信绝大多数软件公司在高并发处理上还是少一些经验的,下面根据我们开发过的高并发项目上总结几点供大家参考:
1、nginx部署负载均衡
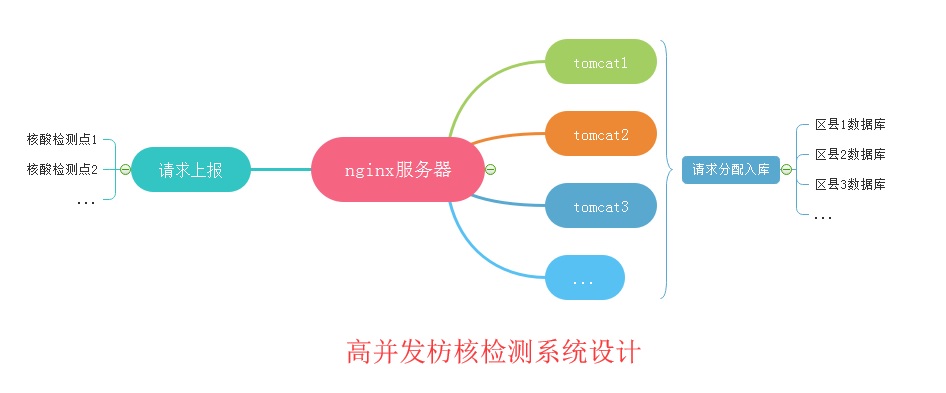
这里我们给nginx部署在一台单独web服务器上,然后通过nginx分发至n台web应用上。
upstream www_balance{ server 192.168.0.101:8080 weight=5; #tomcat1的端口和权重 server 192.168.0.102:8088 weight=5; #tomcat2的端口和权重}
上面配置了两台服务器及tomcat,这个根据你的服务器配置,像我们4核16g内存的服务器,我们一般部署4个tomcat,也就是说假如你有两台应用服务器,可以部署8个tomcat。
在站点的配置上定义
proxy_pass http://www_balance;
2、tomcat的优化
tomcat的默认参数一定要优化!直接copy下面的参数即可
<Connector port="8080" protocol="org.apache.coyote.http11.Http11NioProtocol" connectionTimeout="20000" maxThreads="1000" minSpareThreads="100" maxSpareThreads="300" acceptCount="1000" redirectPort="8443" disableUploadTimeout="true" enableLookups="false" />
3、mysql优化
将以下脚本存为nolimi.sh,然后执行即可
#!/bin/sh
if cat /etc/security/limits.conf | grep "* soft nofile 65535" > /dev/null;then
echo ""
else
echo "* soft nofile 65535" >> /etc/security/limits.conf
fi
if cat /etc/security/limits.conf | grep "* hard nofile 65535" > /dev/null ;then
echo ""
else
echo "* hard nofile 65535" >> /etc/security/limits.conf
fi
修改mysql配置文件/etc/my.cnf,在[mysqld]标签里添加以下代码:max_connections=3600
以上是常规操作,没有太多要讲的,按上面的配置即可。下面说下核心的部分,业务系统设计:
4、数据库设计
这里比较重要!我们保存核酸检测的信息主要有:
姓名、身份证号、电话、区/县、乡镇、街道/村、小区、单元、房屋编号、核酸检测编码、核酸检测机构、核酸检测结果等。
针对上述字段信息,第一件事,我们要做分库!!记着能做分库的,不要做分表。为了将来灵活扩容,因为分表还是压力在一个库上,而一个库是不能随便拆分至不同服务器的。而分库,我们可根据访问量选择把多个数据库部署在一台服务器上,还是每个数据库各自占用一台服务器。
介于此,我们考虑有两种分库方案:
a、按出生日期年份分库,每一年出生的人的核酸信息记录在一个数据库上,我们粗略统计一下1000万人口的城市,按80岁年龄段,,平均一年一个库的话,这样一个数据库库记录在十几万左右。这个数据量完全够mysql快速响应的。如果觉得库太多,也可以分段,每10年的分在一个库中也可以。
b、按行政区分,可以按街道或区、县来创建分库。道理同上。
另一个重要的事,以上数据库绝对不要加索引(本次的核酸检测码作主键),目的就是为了保证最快的写库操作,也就是为了确保现场核酸登记时,高并发下的快速响应,只有响应够快,nginx,tomcat理论上的配置并发数才会达到最高。这个不难理解,比如nginx有10000的并发,同一时刻来了10000人同时提交检测,nginx把请求交给各个tomcat,如果30秒内都没有响应完成,那么第一时刻来的请求到后面30s内再来的请求就只能排队等候了。
相应如果tomcat都能在1秒内处理完请求,那么随着请求的建立和释放动态的过程,一直会有空闲的连接数响应新的检测请求就可以了。因为现场场景中,医务人同每次扫码两个人间隔时间起码也得有2-3秒的间隔,关键点来了,我们java应用每一次响应的用时理论上只要小于现场医务人员两次扫码确认的时间即可达到良好的检测体验。大概情况 就是这样:
10000人并发提交-》nginx响应分发-》n个tomcat各自接收到大概100左右请求->花费1s时间处理完毕->nginx连接释放-》过1-2s(空闲10000连接)-》新到10000请求,以此类推....
有人讲,上面道理不假,如何保证1s处理完,这个不难,参考上面的数据库设计(分库)100%可以做到!
最后有人说nginx10000并发数够用吗?这里nginx可是单点啊,本文讲的是1000万人口,并不是1000万并发,我们算一下1000万人口的城市,最多有1万个核酸检测点,假如最巧合的情况,这1万的检测点同时上报采样,那最极端的情况也就是10000个并发连接。所以nginx这个配置是足够使用了。那为啥有的系统配置了nginx2w的并发数,也不行呢?原因就在于应用层tomcat中的java应用响应太慢了,请求时间占用太久,把连接数都占用了。所以确保应用系统快速入库响应的能力是最根本的解决方法。
还有人问,java中分库的代码,有参数吗?我们也有:),我们多年用自己开发的框架(非spring),一直做多库分库操作。
以下参考:
package totcms.db;
import java.util.Hashtable;
import javax.naming.Context;
import javax.naming.InitialContext;
import javax.sql.DataSource;
import org.apache.commons.logging.Log;
import org.apache.commons.logging.LogFactory;
/**
*
* @ totcms多库
*/
public class DBMap {
private static Hashtable ds = new Hashtable();
private static Log log = LogFactory.getLog(DBMap.class);
static {
String[] s={"mydb_bid","mydb_sale","mydb_buy"};//数据源
try {
Context initContext = new InitialContext();
Context envContext = (Context) initContext.lookup("java:/comp/env");
for(int i=0;i<s.length;i++){
ds.put(s[i],(DataSource) envContext.lookup(s[i]));
log.info("DBMap init datasource = " + s[i]);
}
} catch (javax.naming.NamingException e) {
e.printStackTrace();
}
}
public static DataSource getDs(String s){
DataSource d=(DataSource)ds.get(s);
if(d==null){
d=createS(s);
}
return d;
}
public static DataSource createS(String s){
DataSource d=null;
try {
Context initContext = new InitialContext();
Context envContext = (Context) initContext.lookup("java:/comp/env");
ds.remove(s);
d=(DataSource) envContext.lookup(s);
ds.put(s,d);
log.info("createS = " + s);
} catch (javax.naming.NamingException e) {
e.printStackTrace();
}
return d;
}
}
上面代码是数据源的管理
数据库的配置在xml文件中:
<?xml version="1.0" encoding="UTF-8"?>
<Context path="/" reloadable="true">
<Resources cachingAllowed="false"/>
<Resource auth="Container" driverClassName="com.microsoft.sqlserver.jdbc.SQLServerDriver" logAbandoned="true" maxIdle="300" maxTotal="300" maxWaitMillis="-1" name="mydb_bid" password="_" removeAbandoned="true" removeAbandonedTimeout="100" type="javax.sql.DataSource" url="jdbc:sqlserver://192.168.0.101:1433;DatabaseName=aaa;characterEncoding=utf-8" username="aaa"/>
<Resource auth="Container" driverClassName="com.mysql.jdbc.Driver" logAbandoned="true" maxIdle="300" maxTotal="300" maxWaitMillis="-1" name="mydb_sale" password="" removeAbandoned="true" removeAbandonedTimeout="100" type="javax.sql.DataSource" url="jdbc:mysql://192.168.0.102/bbb?useUnicode=true&characterEncoding=UTF-8" username="root"/>
</Context>
细心可以看到,可以同时支持sqlserver,mysql,oracel等各种数据库。
最后还可能有人问到,上面的库没有做索引,那么检测结果的查询怎么办,这个涉及到另一个库设计:读写分离。上面的库主要解决写的问题,至于读,其实好办了,毕竟核酸检测结果的生成是检测机构操作,可以在检测机构更新检测结果的时候,把该条数据读取写到一个做了索引(按身份证号)的核酸结果数据库中供查询使用,这个数据库的设计 可以按月分库,按日分表设计)
最后上个构架图
检测上报
结果查询


 鲁公网安备 37010302000477号
鲁公网安备 37010302000477号